抑郁症与大脑皮层下结构体积之间的共同遗传基础
研究小组师姐推荐了一篇文献,我进行了学习并且将其与目前工作进行结合联系。文献标题为:《Investigating the shared genetic architecture between depression and subcortical volumes》,探索抑郁症与大脑皮层下结构体积之间的共同遗传基础,文章发表在NC上,发表时间为02 September 2024;

标题和作者信息如上:
文章标题为《抑郁症与皮质下体积的共同遗传结构研究》,这个标题对我而言,有两个不太了解1.皮质下体积是什么;2.遗传结构是什么意思?同时对于所有的文献一样,我需要看作者为什么要将两者进行联系,以及如何将二者进行关联的……先从摘要看起:
📘摘要概述
抑郁症是一种广泛且高度遗传的心理疾病;
现有的影像研究已经发现,与抑郁症相关的皮质下结构(皮质下结构解剖结构见下图)体积异常;
但是抑郁症与皮质下体积之间的遗传基础仍然未被充分理解;
研究思路(从 GWAS → MiXeR → cFDR/conjFDR → shared loci):
- 数据:最大规模的抑郁症全基因组关联研究(N = 674,452 例)和 14 种皮层下容积表型(N = 33,224 例)的汇总统计数据;
- 方法:
- 双变量因果混合模型(MiXeR)调查多基因重叠的程度;
- 通过条件/联合 FDR 分析识别共享基因组位点
结果:MiXeR 分析显示:皮层下结构的体积特征与抑郁症共享了相当比例的遗传变异位点,随后使用共轭 FDR 分析共识别出了 44 个明确的共享遗传位点。这些共享位置映射的269个蛋白编码基因表现出特定的发展轨迹,其中55个基因的表达水平与抑郁和皮质下容量相关,30个基因与认知能力和行为症状相关。
意义:这些发现突显了抑郁症和皮质下的体积表型之间的共享遗传结构,丰富了我们对抑郁症神经生物学基础的理解。

Jung, R. E., Ryman, S. G., Vakhtin, A. A., Carrasco, J., Wertz, C., & Flores, R. A. (2014). Subcortical correlates of individual differences in aptitude. PloS one, 9(2), e89425.
尾状核(caudate)、壳核(putamen)、苍白球(globus pallidus)、丘脑(thalamus)、海马体(hippocampus)、杏仁核(amygdala)、伏隔核(nucleus accumbens)
好了**:laughing:**,摘要看完了,之前的问题可以回答了:1.皮质下体积就是皮质下结构的体积;2.遗传结构就是基因位点;3.关联方式就是MiXeR;
现在我又有了问题:1.摘要说与抑郁症相关皮质下结构体积异常,那这个与抑郁症相关是怎么得到的呢?2.就是这个数据库是什么,基因的数据结构是怎么样的?3.就是这个模型是什么运行的,共轭FDR又是什么?
笔者实力较弱,所以还需要先看一下前言与方法:yum:
:open_file_folder:介绍
抑郁症的流行病学调查与行为/认知症状;
皮层下结构与在情绪、记忆和认知功能中发挥关键作用。所以皮层下结构在抑郁症神经生物学上有重要地位;
皮层下区域在抑郁症中展现出了结构上的变化;
在一个涉及 130 万人的超大规模元分析 GWAS中,研究者找到了243个与抑郁症相关的独立风险基因位点(loci)。
抑郁症和皮层下结构的体积性状都表现出高度的遗传性(heritability),并且都具有多基因特征(polygenic nature)。所以有理由推测,两者在影像表现上的一些相似性,可能源自共享的遗传基础(shared genetic basis)
以往研究使用LDSC(连锁不平衡评分回归)和PRS(多基因风险评分)等常见方法来探索抑郁和皮层下结构体积之间的遗传重叠,但结果发现:
- 基因组范围的遗传相关性很弱甚至缺失(即传统方法找不到强的共享信号)。
LDSC 会尝试计算所有 SNP 的效应方向是否一致来推断“两个性状是否遗传相关”。这会导致 正反抵消(加权求和接近于0)
双变量因果混合物模型(MiXeR):其最大优点在于它会考虑所有的遗传变异,而不管这些变异的效应方向是一致还是相反。通过这种方式,MiXeR能够更准确地评估出两种性状背后真正的遗传重叠程度,不会因为效应方向的矛盾而被误导。
cond/conjFDR 分析 (Conditional & Conjunctional False Discovery Rate):通过整合两个全基因组关联研究(GWAS)的数据,互相提供信息,从而“放大”那些微弱但真实的遗传信号。
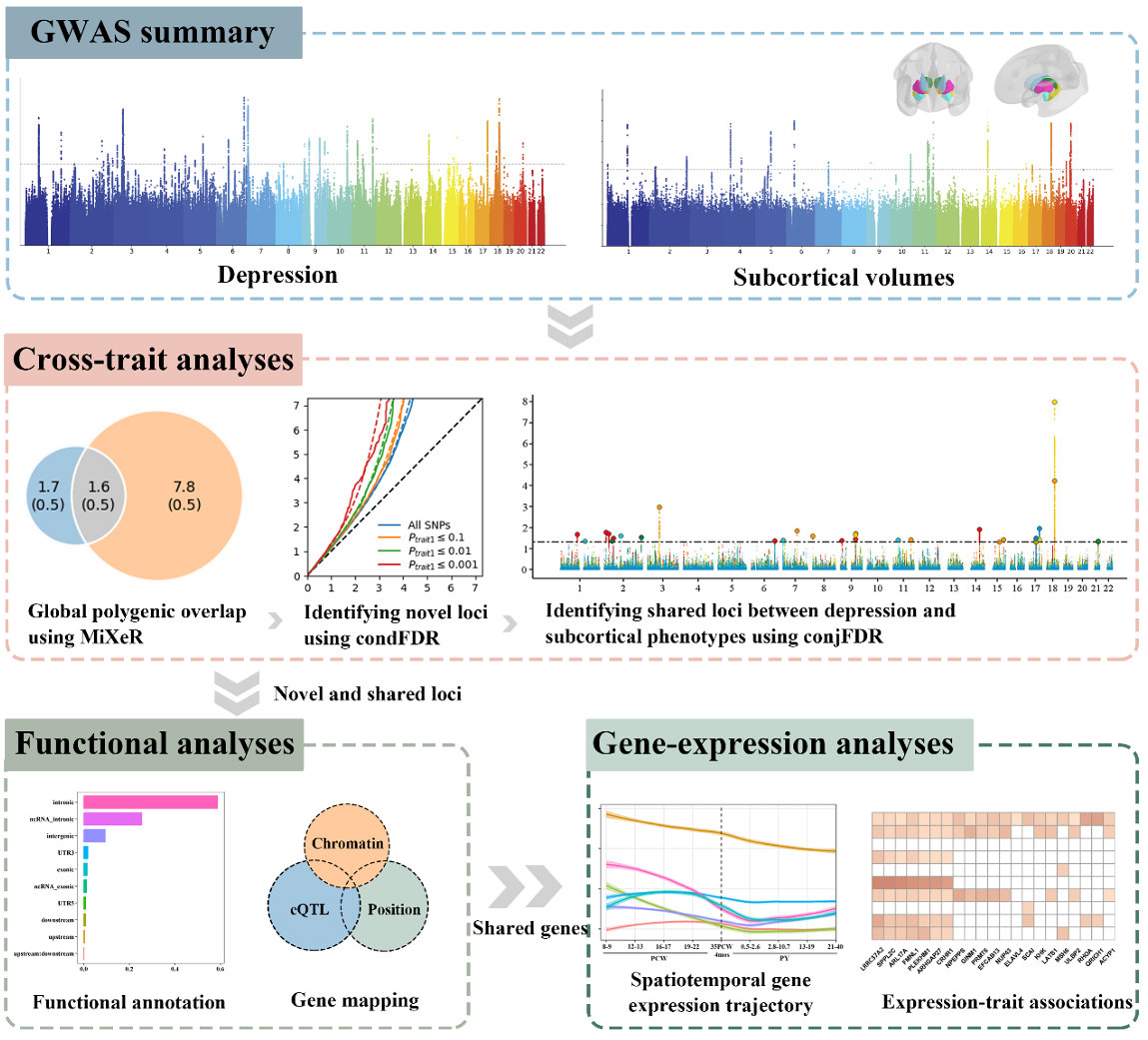
流程设计:研究基于抑郁症和皮层下脑区体积表型的 GWAS 总结统计数据,首先使用 MiXeR 分析两者的多基因重叠情况。随后,使用条件 FDR(condFDR)和联合 FDR(conjFDR)方法识别新的与共享的遗传位点。进一步通过功能注释与基因定位,发现与两种表型相关的共享基因。我们随后分析这些共享基因在不同发育阶段的表达模式,并探索其表达对抑郁症、皮层下结构体积、认知能力和行为症状的潜在影响。跳转到图像注释
结果
Quantification of genetic overlap with MiXeR
🧬 一、单变量 MiXeR 分析(Univariate MiXeR)
✅ 目的:
估算每个性状的:
- SNP 遗传率(SNP heritability)
- 多基因性(Polygenicity,即影响该性状的变异数量)
- 可发现性(Discoverability,即变异能被 GWAS 检测出来的概率)
| 指标 | 抑郁症(Depression) | 皮层下体积(Subcortical Volumes) |
|---|---|---|
| SNP 遗传率 | 0.051(较低) | 0.181 ~ 0.295(较高) |
| 多基因性 | 约 9400 个变异(高) | 939 ~ 3207 个变异(低) |
| 可发现性 | 8.3 × 10⁻⁶(极低) | 1.2 × 10⁻⁴ ~ 3.5 × 10⁻⁴(明显更高) |
| 达到 90% 遗传率所需样本量 | 约 6580 万人 | 约 250 万 ~ 660 万人 |
✅ 解释:
- 抑郁症是一个典型的高度多基因但难以检测的复杂疾病。
- 皮层下体积性状更容易在 GWAS 中检测出显著 SNP(discoverability 更高)。
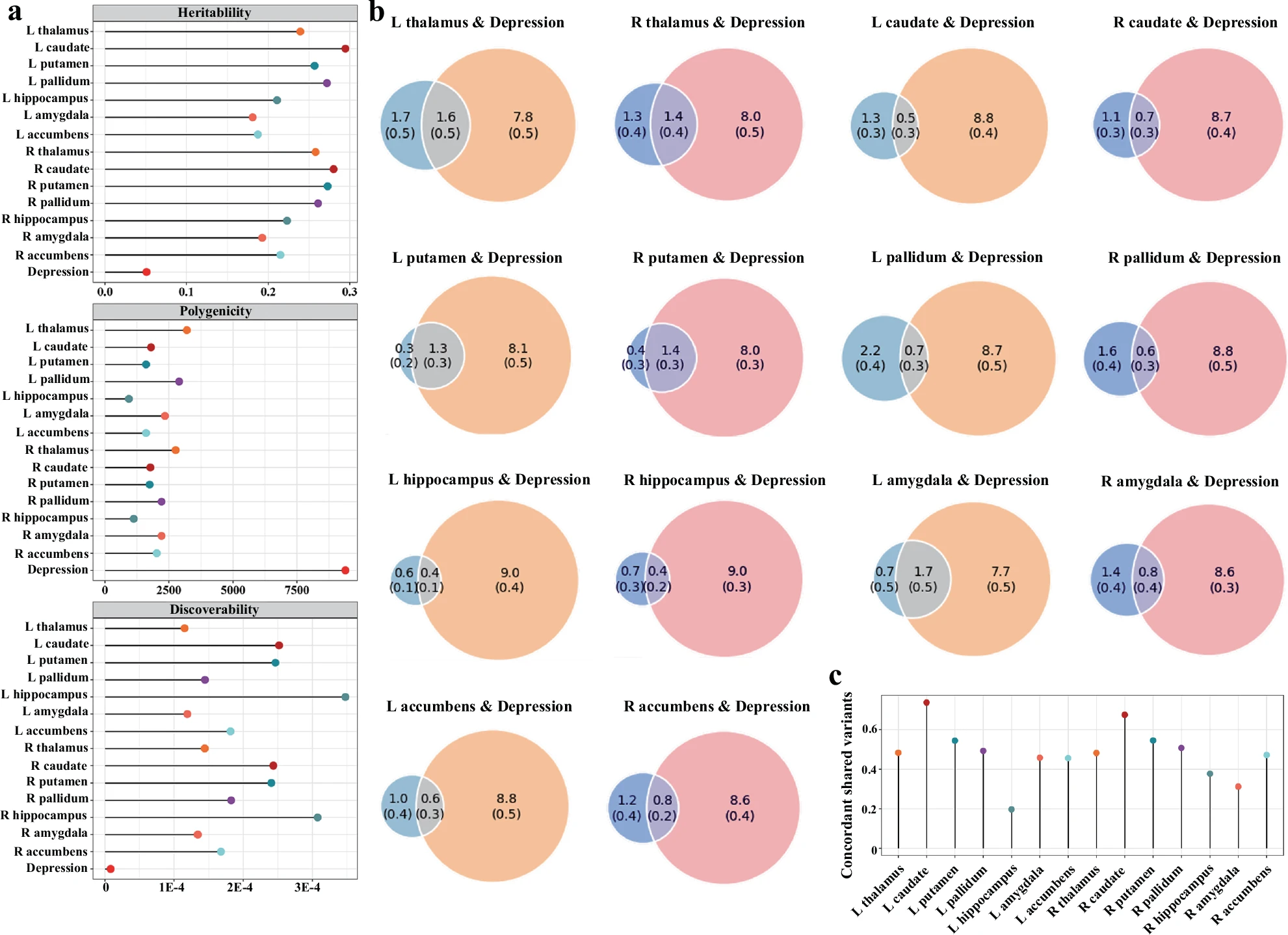
Fig. | Genetic overlap between depression and subcortical volumes using MiXeR.
a Heritability, polygenicity, and discoverability for each phenotype estimated by univariate MiXeR. b Venn diagrams showing MiXeR results of the estimated number of variants shared between depression and subcortical volumes. The numbers of shared and trait-specific variants are shown in thousands with standard errors. The size of the circles reflects the degree of polygenicity. c The points indicate the proportions of shared variants with concordant effect directions. Source data are provided as a Source Data file. L left, R right.
🧬 二、双变量 MiXeR 分析(Bivariate MiXeR)
✅ 目的:
- 评估 抑郁症与各个皮层下脑区体积之间的共享遗传变异数量
- 判断共享变异方向是否一致(即是否协同作用)
🔍 分析流程与标准:
- 14 个皮层下结构中,有 8 个与抑郁症配对后模型拟合良好(AIC 条件满足)
🔍 结果1:共享变异比例
| 指标 | 数值范围 |
|---|---|
| 皮层下体积中与抑郁共享的变异比例 | 23.4% ~ 83.3% |
| 抑郁中与皮层下体积共享的变异比例 | 4.1% ~ 17.9% |
| Dice 系数(共享变异/总变异) | 7.4% ~ 28.5% |
🔎 解读:共享程度不对称,体积特征比抑郁症更容易共享变异;这说明抑郁症中许多变异是特异的,而体积中的变异更泛化。
🔍 结果2:共享变异效应方向分析
- 即:共享变异是朝同一个方向影响两个性状,还是相反方向?
- 结果显示:共享变异中方向一致性从 19.7% 到 73.6% 不等 → 有些脑区与抑郁症是正相关,有些是负相关
| 结构 | 与抑郁相关方向 | 相关系数 (rho) |
|---|---|---|
| 左尾状核(Left Caudate) | 正相关 | 0.634(最大) |
| 双侧壳核(Putamen)、右苍白球(Right Pallidum) | 正相关 | 中等正相关 |
| 左海马(Left Hippocampus) | 负相关 | -0.784(最大) |
🔎 解读:
- 尾状核和壳核等区域可能在与抑郁的易感性或补偿机制有关;
- 海马等区域则可能参与抑郁症的病理性萎缩或功能损伤。
📌 总结一句话:
使用 MiXeR 分析揭示了抑郁症和皮层下脑区体积之间存在不同程度的遗传重叠,但其变异数量、效应方向和生物学机制呈现出高度异质性,说明这两类性状虽有关联,但背后调控机制复杂且不完全重叠。
🧬 Identification of novel and shared genetic loci (cond/conjFDR)
本部分通过 condFDR 和 conjFDR 方法,系统鉴定了抑郁症与皮层下脑区体积表型之间的 新颖和共享遗传位点,揭示其复杂的共遗传结构与潜在功能机制。
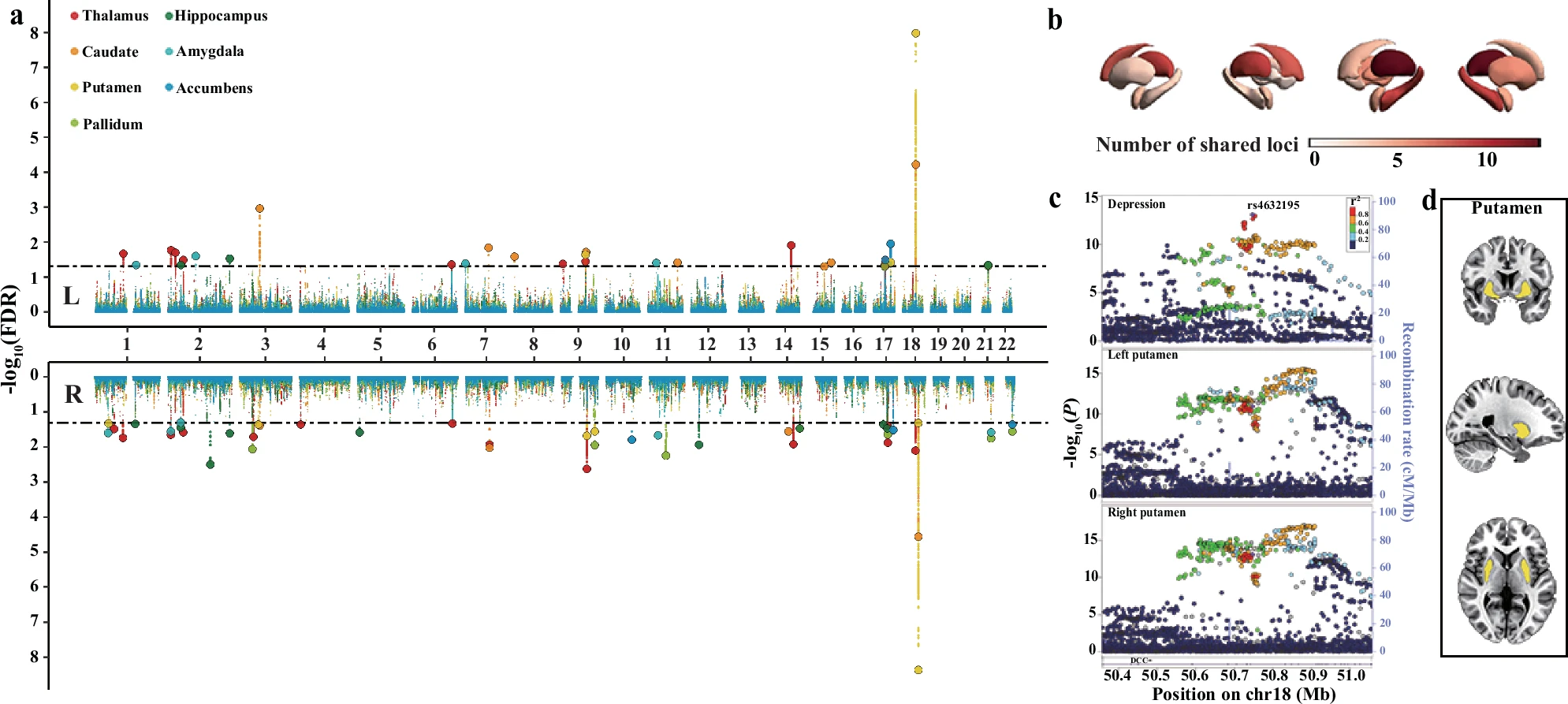
a Manhattan plot showing the associations between depression and subcortical volumes identified by conjFDR. Each point represents a SNP and the point with a larger size and black border indicates a lead SNP. The y-axis represents the -log10-transformed conjFDR values for each SNP, and the x-axis reflects the chromosomal position. The dashed line represents the threshold of conjFDR = 0.05, and the color of the dots represents different phenotypes. b Brain map showing the number of loci of each subcortical structure shared with depression. A color transition from white to red indicates an increasing number of shared loci. c LocusZoom plots of a genomic locus (chr18: 50358109-51055069, lead SNP: rs4632195) showing the regional SNP associations (two-sided -log10(p)) for depression and bilateral putamen volumes from the original GWAS summary data d. MRI slice plot of bilateral putamen volumes. Source data are provided as a Source Data file. Chr chromosome, L left, R right.
🔍 1. 条件 FDR(condFDR)分析结果
| 分析方向 | 发现数量 | 备注 |
|---|---|---|
| 抑郁症(以皮层下结构为条件) | 94 个关联位点(FDR < 0.01) | 包含 7 个新位点 |
| 这些新位点关联的基因 | 14 个 | GO富集分析未显著(因基因数过少) |
| 皮层下体积(以抑郁症为条件) | 410 个位点 | 分布于 7 个脑区,详见下表 |
| 汇总所有皮层下表型后 | 233 个独立位点 | 含 13 个新位点,映射到 57 个基因 |
| GO 富集结果 | 与 GABA能神经元发育 和 嘧啶核苷合成过程 相关(q=4.0E-2) |
➤ 410 loci 在各脑区的分布:
| 脑区 | 左侧(N) | 右侧(N) |
|---|---|---|
| 丘脑 (Thalamus) | 22 | 28 |
| 尾状核 (Caudate) | 43 | 41 |
| 壳核 (Putamen) | 44 | 43 |
| 苍白球 (Pallidum) | 20 | 35 |
| 海马 (Hippocampus) | 25 | 33 |
| 杏仁核 (Amygdala) | 19 | 12 |
| 伏隔核 (Accumbens) | 25 | 20 |
🧬 2. 联合 FDR(conjFDR)分析结果
| 指标 | 数值 |
|---|---|
| 发现共享位点总数 | 77 个(conjFDR < 0.05) |
| 涉及的皮层下脑区 | 7 个 |
| 映射至的 distinct 共享位点 | 44 个 |
| 其中新发现的:— 对抑郁症新— 对皮层下表型新— 双方皆新 | 7 个17 个2 个 |
➤ 77 shared loci 在各脑区的分布:
| 脑区 | 左侧(N) | 右侧(N) |
|---|---|---|
| 丘脑 | 9 | 13 |
| 尾状核 | 8 | 4 |
| 壳核 | 3 | 6 |
| 苍白球 | 2 | 6 |
| 海马 | 3 | 9 |
| 杏仁核 | 4 | 5 |
| 伏隔核 | 2 | 3 |
⭐ 3. 最显著共享位点:
- 位置:chr18: 50358109–51055069
- SNP:rs4632195
- 对应脑区:双侧壳核(putamen)
- conjFDR:
- 左侧 putamen:1.0E−8
- 右侧 putamen:4.3E−9
🧬 4. 功能注释结果(44 个共享位点)
| 内容 | 发现 |
|---|---|
| 映射基因数量 | 269 个 protein-coding genes |
| GO 富集条目 | 42 个(q < 0.05) |
| 代表性生物过程 | 神经投射发育、轴突发生调控、中枢神经系统发育 |
🧪 5. SNP 功能特征分析:
| 类别 | 百分比 | 示例或说明 |
|---|---|---|
| 位于内含子区 | 58.7% | 普遍情况 |
| 位于 lncRNA 内含子区 | 25.4% | |
| 非同义突变 SNP | 18 个 | 可能有直接功能 |
| CADD > 12.37(潜在致病) | 3.7% | 最高为 rs62618693(CADD = 31) |
| RegulomeDB < 3(调控功能) | 10.3% | |
| 开放染色质区域(ChromHMM < 8) | 90.1% |
🧪 6. 效应异质性分析(Cochran’s Q test)
| 分析对象 | 结果 |
|---|---|
| 所有 44 个 SNP 在全部 14 个脑区中 | 有 30 个存在显著效应异质性(p < 0.05) |
| 其余 14 个 | 无显著异质性 |
| 双侧脑区(如左右尾状核) | 仅伏隔核中一对有异质性 |
| 异质性最多的脑区对 | 左尾状核 – 右海马(20 个 SNP) |
| 异质性最少的脑区对 | 左海马 – 左杏仁核(2 个 SNP) |

a Manhattan plot showing the associations between depression and subcortical volumes identified by conjFDR. Each point represents a SNP and the point with a larger size and black border indicates a lead SNP. The y-axis represents the -log10-transformed conjFDR values for each SNP, and the x-axis reflects the chromosomal position. The dashed line represents the threshold of conjFDR = 0.05, and the color of the dots represents different phenotypes. b Brain map showing the number of loci of each subcortical structure shared with depression. A color transition from white to red indicates an increasing number of shared loci. c LocusZoom plots of a genomic locus (chr18: 50358109-51055069, lead SNP: rs4632195) showing the regional SNP associations (two-sided -log10(p)) for depression and bilateral putamen volumes from the original GWAS summary data d. MRI slice plot of bilateral putamen volumes. Source data are provided as a Source Data file. Chr chromosome, L left, R right.
Developmental trajectories of shared genes
🧠 使用数据:
- 数据来源:PsychENCODE 项目
- 内容类型:人类大脑 不同发育阶段 和 不同脑区 的基因表达数据
利用了PsychENCODE35的时空大脑基因表达数据,作者研究了抑郁症与皮层下结构共有基因的发育表达轨迹。如Fig. 3c
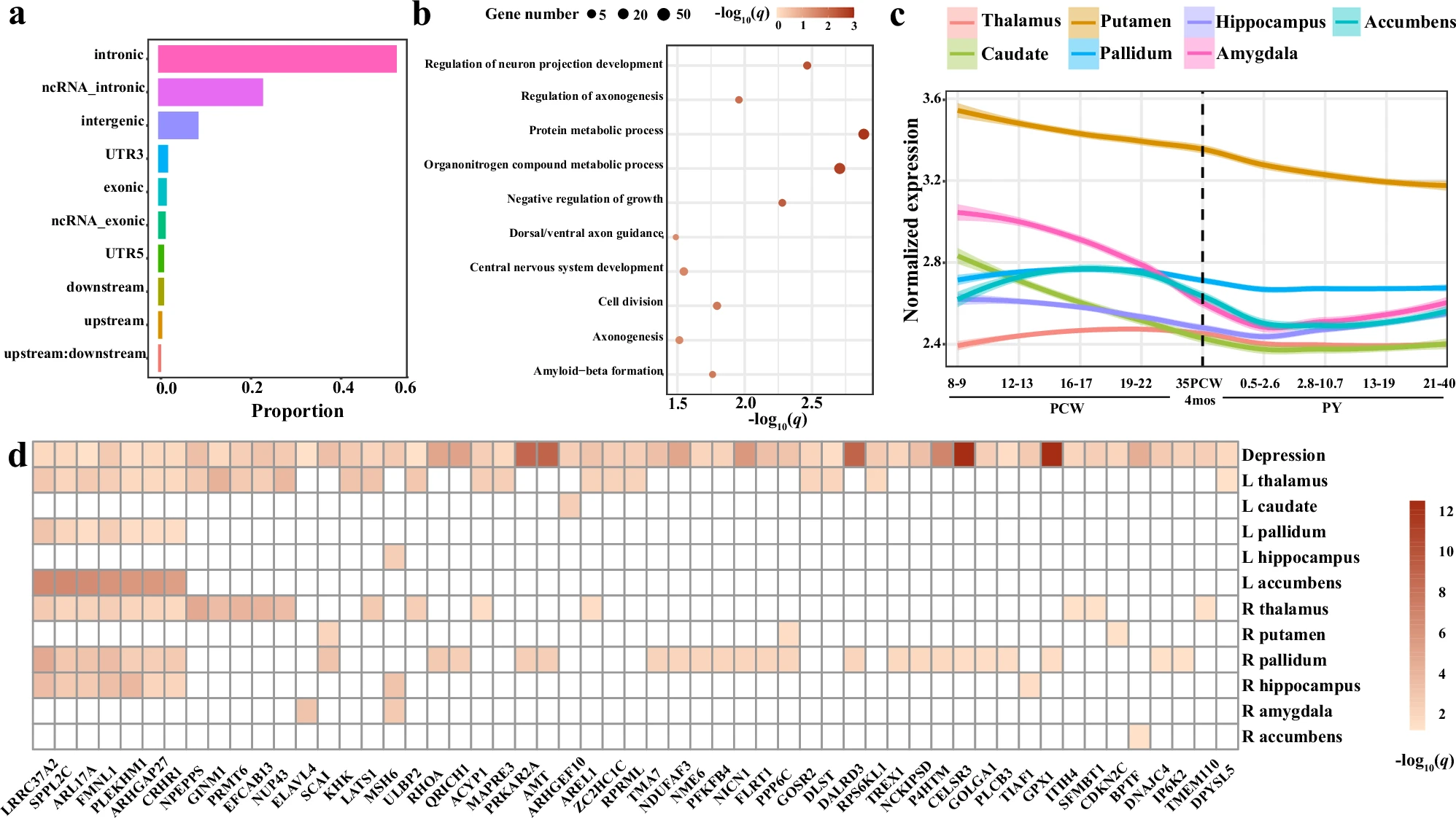
| 窗口 | 阶段 | 具体时间 |
|---|---|---|
| W1 | 早期胎儿期 | 胎龄 8–9 周(PCW 8–9) |
| W3–4 | 中期胎儿期 | 胎龄 16–22 周(PCW 16–22) |
| W6 | 婴幼儿早期 | 生后 0.5–2.6 岁(PY 0.5–2.6) |
各脑区共享基因表达趋势
| 脑区 | 表达轨迹 | 高峰期 | 后续变化 |
|---|---|---|---|
| 伏隔核(accumbens) 苍白球(pallidum) 丘脑(thalamus) | 表达模式相似:中胎期达到高峰 | PCW 16–22(W3–4) | 稍微下降后,在 W6(PY 0.5–2.6)进入平台期 |
| 海马(hippocampus) 杏仁核(amygdala) 尾状核(caudate) | 早孕期即达高峰,然后持续下降 | PCW 8–9(W1) | 表达量持续下降,至婴幼儿期(PY 0.5–2.6)达到最低 |
| 壳核(putamen) | 也是早期高峰 | PCW 8–9(W1) | 持续下降,与上面类似但无平台期 |
🧠Expression-trait associations(基因表达与性状的关联分析)
🔬 工具与目的:
- 使用 S-MultiXcan 工具对每一对“抑郁症–皮层下体积表型”共享基因性状进行分析。
- 目标:研究这些 共享基因在大脑中的表达水平,是否同时与:
- 抑郁症;
- 对应的皮层下脑区体积;
存在显著统计关联。
📊 分析结果概览:
- 共发现 137 个显著关联(FDR 校正后);
- 涉及 71 个共享基因 与不同的皮层下体积;
- 其中有 55 个基因 同时也与抑郁症显著相关。
:link:Cognitive and behavioral associations
研究目的:抑郁症–皮层下体积表型”共享基因是否进一步影响抑郁症相关的认知和行为性状;
共有 8 种认知能力和 15 种行为症状的数据
🧬 分析背景:
- 分析对象:55个“共享基因”
- 即那些 同时与抑郁症和至少一个皮层下脑区体积相关的基因;
- 工具:S-MultiXcan;
- 目标:探究这些基因的大脑表达水平是否与:
- 认知能力(cognitive abilities);
- 行为症状(behavioral symptoms);
存在统计学显著关联。
📊 主要发现:
| 分析方向 | 显著关联数 | 涉及基因数 |
|---|---|---|
| 🧠 基因表达 vs. 行为症状 | 30 个显著关联 | 30 个基因 |
| 🧠 基因表达 vs. 认知能力 | 59 个显著关联 | 30 个基因 |
👉 说明:有些基因同时关联了多个认知与行为指标。
此外,文章还观察到同一基因在不同皮层下区域的表达水平与不同的认知能力或行为症状相关(图 4b,c)。
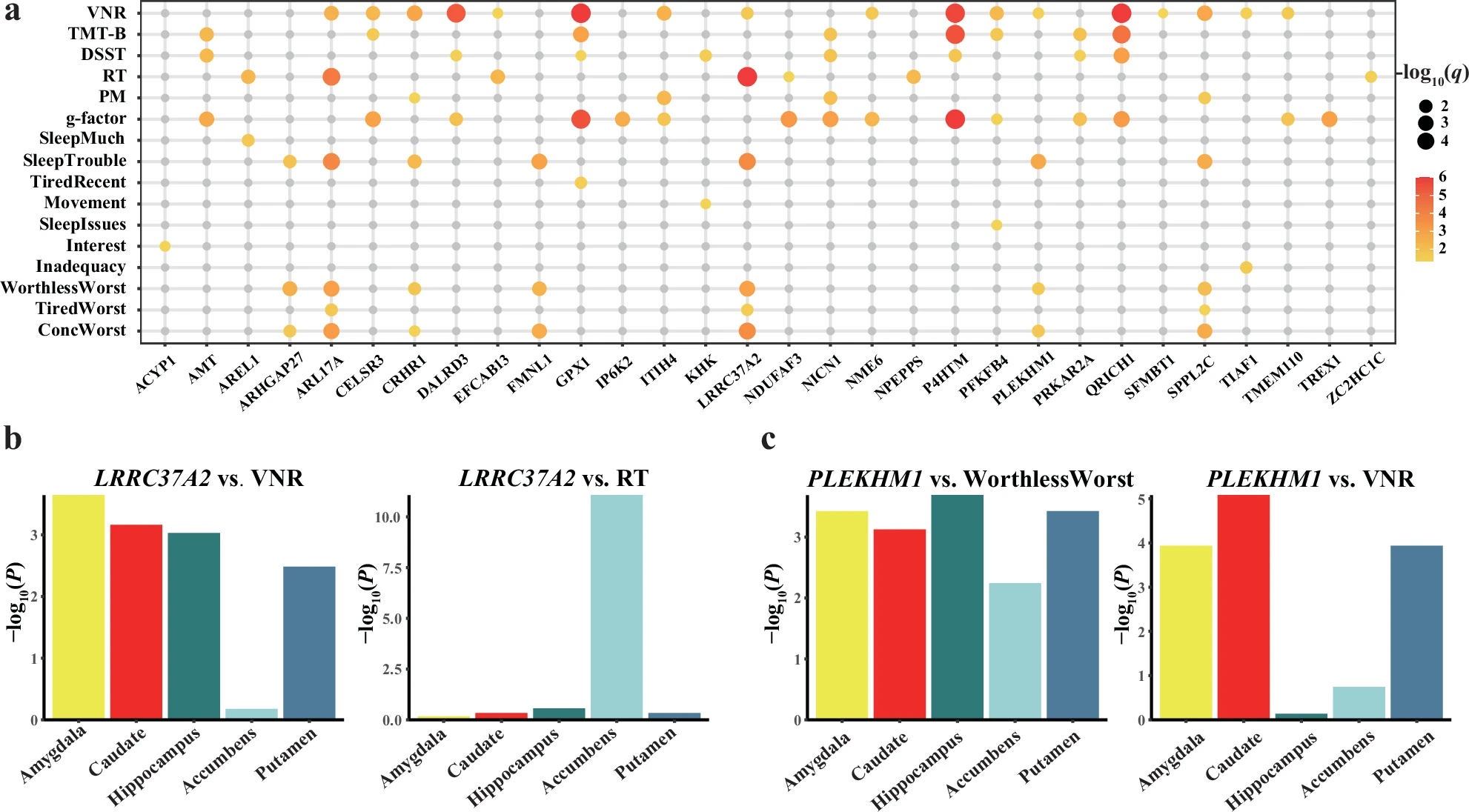
Fig. 4 | Associations of the expression of shared genes in the brain with cognitive abilities and behavioral symptoms. a Bubble plot showing significant expression-trait associations (two-sided q < 0.05, FDR corrected). The x-axis shows the shared genes, and the y-axis shows cognitive abilities and behavioral symptoms. The size and color of the bubbles demonstrate the significance level. The gray bubbles represent non-significant associations. b The bar plots show the significance level (uncorrected two-sided p-values) of the expression of LRRC37A2 in five subcortical structures with the VNR and RT. c The bar plots show the significance level (uncorrected two-sided p-values) of PLEKHM1 expression in five subcortical structures associated with feelings of worthlessness during the worst period of depression and VNR. Source data are provided as a Source Data file. ConcWorst difficulty concentrating during worst depression, DSST digit symbol substitution task, Inadequacy recent feelings of inadequacy, Interest recent lack of interest or pleasure in doing things, Movement recent changes in speed/amount of moving or speaking, PM pairs matching, RT reaction time, SleepIssues trouble falling or staying asleep, or sleeping too much, SleepMuch sleeping too much, SleepTrouble trouble falling asleep, TiredRecent recent feelings of tiredness or low energy, TiredWorst feelings of tiredness during the worst episode of depression, TMT-B trail making test B, VNR verbal–numerical reasoning, WorthlessWorst feelings of worthlessness during worst period of depression.
⭐️ 多重影响基因(影响广泛的代表性基因):
5 个基因 与5种以上认知/行为指标显著相关
附录

🧭 整体流程结构
该图分为四个主要模块,从上到下依次为:
- GWAS summary
- Cross-trait analyses
- Functional analyses
- Gene-expression analyses
中间用虚线箭头表示数据或信息的流向。
🟦 1. GWAS Summary
图中内容:
- 左侧是抑郁症(Depression)的曼哈顿图
- 右侧是皮层下脑区体积(Subcortical volumes)的曼哈顿图
- 旁边展示了大脑皮层下结构的3D可视化
含义:
- 使用了两个独立的GWAS数据集,分别研究抑郁症和皮层下脑区体积的遗传基础。
🟥 2. Cross-trait analyses
这一部分核心是识别两个性状之间的共享遗传位点。
(1)Global polygenic overlap using MiXeR
- 使用 MiXeR 方法,评估两个表型的全局多基因重叠程度
- Venn图展示了每个表型的有效SNP数量及其重叠(蓝色1.7表示仅在抑郁中显著,橙色7.8仅在体积中显著,重叠为1.6)
(2)Identifying novel loci using condFDR
- 显示的是一个 Q-Q 图,用条件 FDR 方法在一个性状上利用另一个性状信息来提升新SNP的发现效率
(3)Identifying shared loci using conjFDR
- 曼哈顿图中展示了通过 conjFDR 方法发现的共享遗传位点,即对两个表型都有显著影响的SNP
🟩 3. Functional analyses
这一部分聚焦于对共享位点的生物学解释
(1)Functional annotation
- 条形图显示了共享SNP在不同功能区域(如启动子、调控区、非编码区等)的分布频率
(2)Gene mapping
- 使用多种策略(染色质相互作用、表达QTL、物理位置)将显著SNP映射到潜在的基因上
🟫 4. Gene-expression analyses
进一步分析这些共享基因在不同阶段、不同表型中的表现。
(1)Spatiotemporal gene expression trajectory
- 左侧图展示了不同时间点(出生前后)脑区中共享基因的表达变化轨迹
- 横轴是发育时间,纵轴是表达水平,不同颜色表示不同脑区
(2)Expression-trait associations
- 右侧热图展示了基因表达与多个表型(如抑郁、体积、认知等)的相关性
- 越深的颜色表示相关性越强
✅ 总结一句话
这张图从原始GWAS数据出发,结合统计分析(MiXeR、FDR)+功能注释+表达分析,层层推进,最终识别出与抑郁症和皮层下脑区体积均相关的关键基因,并探索其在不同生命阶段的表达模式及其对认知和行为的影响。
以下是文中提到的关键指标解释表,适用于阅读理解 MiXeR 分析报告或科研论文:
🧪 MiXeR 分析中常见指标解释表
| 指标名称 | 英文名 | 含义解释 | 解读要点 |
|---|---|---|---|
| SNP 遗传率 | SNP heritability | 某一性状中可以被SNP 解释的遗传变异比例(属于全遗传率的一部分) | 数值越高,说明这个性状更容易被基因解释,GWAS 分析更有可能找到显著结果 |
| 多基因性 | Polygenicity | 影响该性状的有效变异(SNP)数量 | 越高表示该性状由大量小效应变异控制(典型如抑郁症),很难用单一或少量基因解释 |
| 可发现性 | Discoverability | 某性状中的遗传变异在 GWAS 中被发现的可能性;数学上等价于平均效应平方 | 数值越大表示 SNP 效应越大、易于检测;越小表示 GWAS 要更大样本量才能发现它 |
| 样本量估计 | Required sample size for 90% heritability | 达到 90% SNP 遗传率所需的 GWAS 样本规模 | 抑郁症需要更大的样本才能找到其相关变异(例如需要上千万样本) |
| 共享变异比例 | Proportion of shared variants | 两个性状之间共享的有效遗传变异的比例 | 比例不对称是正常的,比如 A 性状与 B 性状共享一部分,但反过来不一定一样多 |
| Dice 系数 | Dice coefficient |  衡量两个性状遗传重叠的程度 衡量两个性状遗传重叠的程度 |
值越高,表示两个性状遗传基础越相似 |
| 一致效应比例 | Proportion of concordant effects | 在共享变异中,SNP 对两个性状方向**相同(均为正或均为负)**的比例 | 可以揭示共享变异是否支持类似或相反的生物作用机制 |
| 相关系数 | ρ(rho) | 两个性状共享变异的效应方向之间的相关性(如尾状核与抑郁症的正相关) | 正值表示两性状朝相同方向变化;负值表示反方向变化 |
🧩 举例补充:
- 抑郁症 SNP heritability = 0.051 → 说明基因只能解释约 5.1% 的变异,说明其受环境和复杂交互影响大。
- 抑郁的 polygenicity = 9400 → 高度多基因特征,许多小效应 SNP。
- discoverability = 8.3E-6 → 说明要非常大的样本量才能检测到这些变异。
- Dice = 28.5%(比如与尾状核) → 说明两性状在该脑区上有相对较强的遗传重叠。