精神分裂症多基因风险相关多模态MRI额颞叶网络的推导和应用

这篇论文是我目前在做的工作的参考,因为我近来在用另一批数据套用这篇文章的方法,思路以及算法有些模糊了,所以重新整理一下。文章发表在Nature Communication,原标题为《Derivation and utility of schizophrenia polygenic risk associated multimodal MRI frontotemporal network》,发表时间是2022年八月。
摘要
精神分裂症(SZ)是一种高度遗传的精神疾病,其特征是广泛的大脑功能和结构异常。然而,之前的 MRI 与多基因风险之间的关联研究大多是基于 ROI 的单一模态分析,而不是识别基于大脑的多模态预测生物标志物。根据英国生物银行数据集中健康白人的精神分裂症多基因风险评分 (PRS)( N = 22,459),我们发现了一种与 PRS 相关的稳健大脑模式,其灰质体积较小,额颞叶皮质功能激活减少,这将精神分裂症与精神分裂症区分开来。控制准确度为 >83%,并预测 4 个独立精神分裂症队列的认知和症状。进一步的多种疾病比较表明,这些确定的额颞叶改变在精神分裂症和精神分裂情感患者中最为严重,在双相情感障碍中较轻,并且与自闭症、抑郁症和注意力缺陷多动障碍的对照者没有区别。这些发现表明,所识别出的与多基因风险评分(PRS)相关的多模态前颞网络具有作为精神分裂症特异性的跨诊断基因介导的脑部生物标志物的潜力。
- 这里有几个问题
- 多模态预测和单模态预测相比有什么优势?
- 为什么要将多基因风险与MRI关联,他们比不关联又有什么优势呢?
- 在我的工作中,我将IL-6作为介导物,如何将他们关联起来。
前言概述
背景介绍
- 精神分裂症与遗传因素-基因风险分数(PRS)关联;
- 精神分裂症与大脑结构有关,部分大脑结构改变与认知有关;
- SZ_PRS与MRI的结构和功能都有关联(啥叫关联?),可能导致心理功能与临床症状的改变。将PRS与MRI进行结合,可以从遗传和脑表型两个角度对精神分裂症相关的潜在的病理生理过程提供互补的见解。
- 目前尽管有报道称精神分裂症患者的大脑异常与 PRS 有关,但研究只关注了一种成像模式,并使用了基于感兴趣区(ROI)的简单相关分析。
- 尚未使用全脑多模态 MRI 融合来识别 PRS 相关模式,包括使用机器学习方法来评估其生物标记物特性。更具体地说,目前还没有针对 SZ 和健康对照(HC)分类的 PRS-多模态联合脑成像研究,也没有利用这些变量预测认知或症状的研究。
- 如果成功,本文提出的方法将是朝着使用成像遗传数据作为潜在生物标记物以协助临床医生进行鉴别诊断和预测相关临床结果迈出的重要一步。
文章思路
研究旨在通过关联精神分裂症的多基因风险评分 (PRS-SZ) 与大脑结构和功能的特定协变模式,来识别精神分裂症的脑影像标记。
研究人员假设:
- PRS-SZ 与一种独特的多模态(静息态功能磁共振成像和结构磁共振成像)大脑模式相关。
- 这种大脑模式能够准确区分精神分裂症患者 (SZ) 和健康对照组 (HC)。
- 这种模式能够预测精神分裂症患者的重要临床指标。
为实现这一目标,该研究包括了几个步骤:
- 发现阶段: 他们使用了来自英国生物样本库 (UK Biobank) 的大规模数据集(N = 22,459名健康白人),基于低频振幅分数 (fALFF) 和灰质体积 (GMV) 来识别与PRS相关的脑模式。
- 内部验证: 随后,在英国生物样本库内部,使用不同的人群样本和变化的遗传风险评分阈值对这种已识别的模式进行了验证。
- 外部验证: 在四个独立(不同于英国生物样本库)的精神分裂症队列中,测试了这种与PRS相关的脑模式对精神分裂症患者进行分类以及预测认知/症状结果的能力。
- 特异性验证: 研究人员还调查了这种脑模式是否为精神分裂症所特有,或者是否也存在于其他精神疾病中。
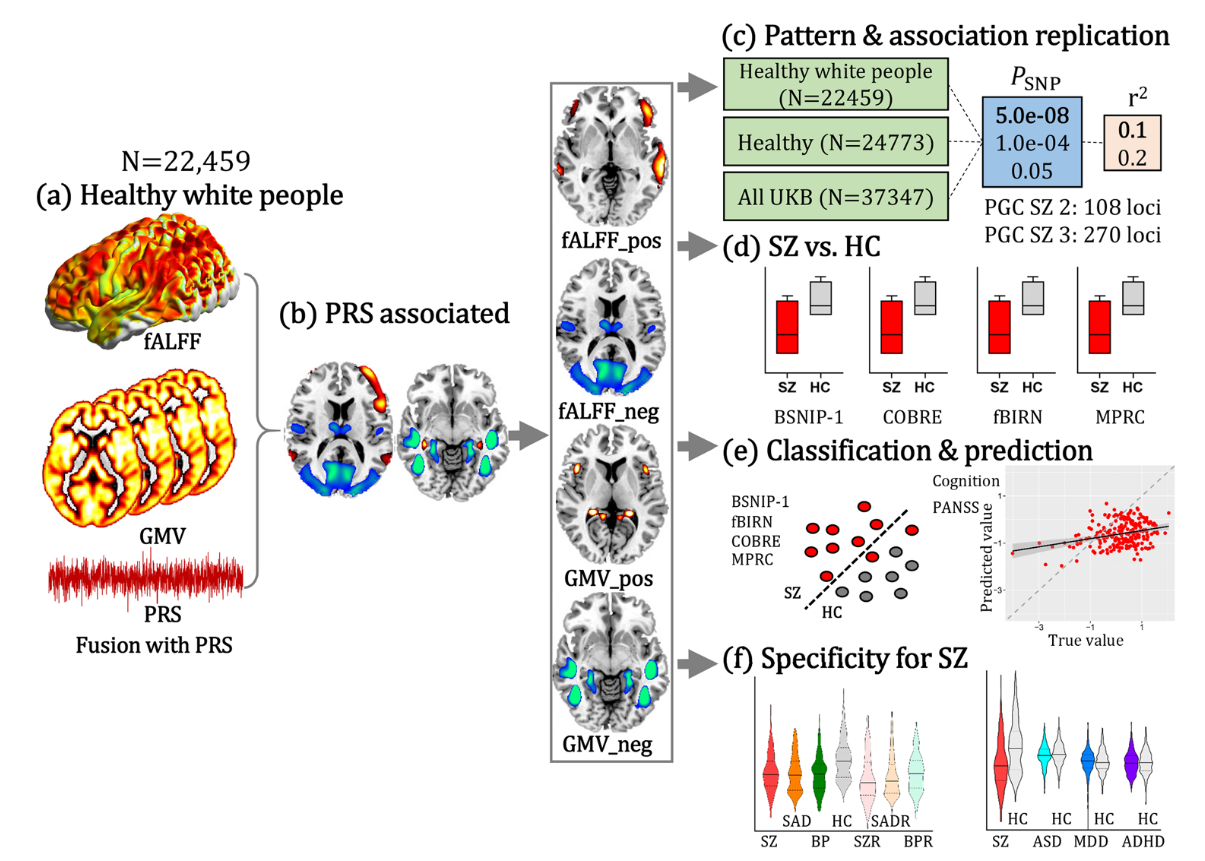
(a) SZ PRS was used as a reference to guide fALFF + GMV fusion to identify PRS-associated multimodal networks. (b) PRS-associated networks were separated as positive (Z > 0) and negative (Z < 0) brain regions based on the Z-scored brain maps, resulting in 4 features for the following analysis. (c) The same fusion with PRS analysis was performed on healthy white people, healthy subjects, and all the available subjects within UKB with PRS thresholded at PSNP = 5.0e−08, 1.0e−04 and 0.05, and pruned at r2 < 0.1 and 0.2, respectively. (d) Group differences between SZ and HC of the identified PRS-associated features were tested across 4 independent SZ cohorts (including BSNIP-1, COBRE, fBIRN and MPRC). (e) Linear SVM was used to classify SZ and HC across 4 independent SZ cohorts based on PRSassociated features (including BSNIP-1, COBRE, fBIRN and MPRC); multiple linear regression was performed to predict cognition and symptom for SZ across 3 independent cohorts (BSNIP-1, COBRE, and fBIRN). MPRC data were not included in the prediction analysis since the related clinical measures were not available. (f) Group differences between SZ and other psychosis subjects and their relatives, and between other psychiatric disorders (ASD, MDD, ADHD) and HC were tested. UKB (UK Biobank, N = 37,347); BSNIP-1 (Bipolar and Schizophrenia Network for Intermediate Phenotypes), SZ: N = 178; HC: N = 220; COBRE (Center for Biomedical Research Excellence), SZ: N = 100; HC: N = 90; fBIRN (Functional Imaging Biomedical Informatics Research Network), SZ: N = 164; HC: N = 157; MPRC (Maryland Psychiatric Research Center), SZ: N = 164; HC: N = 157; ASD autism spectrum disorder, MDD major depressive disorder, ADHD attention-deficit/hyperactivity disorder, SVM support vector machine.
最终目标是,通过整合来自大规模英国生物样本库样本和四个独立精神分裂症队列的多基因风险评分数据与多模态MRI特征,建立一个与精神分裂症遗传风险相关的、更稳健的功能-结构MRI特征。
结果
SZ PRS-associated multimodal brain network
这段文字描述了研究者如何识别与精神分裂症多基因风险评分 (PRS-SZ) 相关的多模态(fALFF + GMV)大脑网络模式。
主要内容如下:
PRS-SZ的计算: 研究者基于精神基因组学联盟 (PGC SZ 2) 的108个风险位点计算了精神分裂症的PRS,并设定了特定的统计学阈值 (PSNP < 5.0e−08, r2 < 0.1)。
数据预处理: 在进行融合分析前,从fALFF(低频振幅分数)和GMV(灰质体积)特征矩阵中回归剔除了头动、研究地点、性别和年龄的影响。
发现队列与方法: 使用英国生物样本库 (UKB) 中22,459名健康白人的数据,通过将PRS与fALFF和GMV数据进行融合分析,以识别与PRS相关的多模态模式。
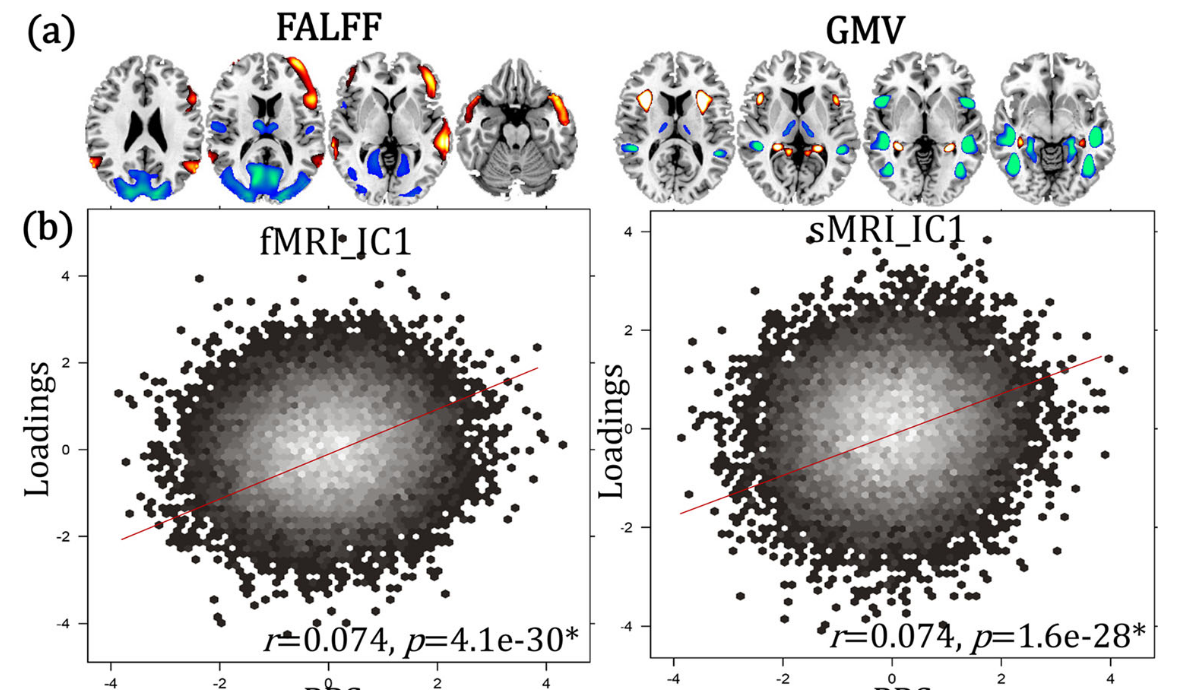
核心发现: 识别出一个与PRS呈正相关的联合成分。这个成分与PRS的fALFF相关性 r = 0.074 (p = 4.1e−30),与GMV的相关性 r = 0.074 (p = 1.6e−28),这些结果都经过了多重比较校正 (FDR)。
效应量与统计功效: 尽管该模式解释的方差小于1%(与以往基于ROI的单模态分析结果一致),但统计功效非常高 (1 − β = 1)。
解释的方差百分比计算方法就是将该联合成分与PRS-SZ之间的相关系数r (0.074) 进行平方,得到R²。
对比分析的局限性: 研究者指出,直接计算PRS与全脑体素水平MRI特征之间的相关性非常弱(fALFF最大绝对相关性r仅为0.03,GMV为0.028)。同样,基于脑图谱提取的平均值与PRS的相关性所解释的方差也小于1%。这凸显了融合分析方法的优势。
“直接计算PRS与全脑体素水平MRI特征之间的相关性非常弱”: 研究者除了使用复杂的“融合分析”方法外,还尝试了更简单直接的方法。一种是逐个体素(voxel,可以理解为MRI图像中的小立方体单元)地计算其fALFF或GMV值与PRS-SZ之间的相关性。结果发现,这种直接的相关性非常微弱。对于fALFF,在全脑数万个体素中,相关性系数r的最大绝对值也只有0.03;对于GMV,也只有0.028。这些r值接近于0,表明几乎没有线性关系。
“同样,基于脑图谱提取的平均值与PRS的相关性所解释的方差也小于1%”: 另一种简单方法是,不看单个体素,而是根据已有的脑图谱(将大脑划分为不同区域的模板)计算每个脑区的平均fALFF或GMV值,再看这些平均值与PRS-SZ的相关性。结果是,这种方法解释的PRS-SZ方差也小于1%,与融合分析得到的整体模式的解释力相似(但融合分析找到了一个特定的“模式”,而这里只是看单个区域平均值)。
“这凸显了融合分析方法的优势”: 这句话是结论。因为上述两种更直接、更简单的方法(逐体素分析、基于图谱的区域平均值分析)得到的相关性要么极低,要么解释力与融合分析方法相仿但未能揭示多模态的“协变模式”。而研究中采用的“融合分析”方法(原文提到“fusion with PRS was performed to identify PRS-associated fALFF + GMV multimodal pattern”)能够同时考虑fALFF和GMV两种模态的数据,并识别出它们如何“共同变化”以与PRS-SZ关联。尽管其解释的总方差也不高,但它能成功识别出一个统计上非常显著的、结构化的、多模态的“联合成分”或“模式”。这表明,精神分裂症的遗传风险可能不是简单地与单个脑区的孤立特征相关,而是与跨多个脑区、跨多种模态特征的复杂组合模式相关,而这种复杂模式需要更高级的分析方法(如融合分析)才能捕捉到。
PRS相关模式的具体描述:
- fALFF表现: 在额中下回 (MIFC)、颞上中回 (SMTC) 与PRS呈正相关(红色区域);在丘脑、后扣带皮层 (PCC)、枕中回 (MOC) 和舌回 (LG) 与PRS呈负相关(蓝色区域)。
- GMV表现: 在前脑岛和海马区与PRS呈正相关;在中脑岛、颞上中下回 (SMITC)、梭状回 (FG) 和海马旁回与PRS呈负相关。
更多细节: 具体的脑区信息(Talairach标签)在补充表2中有详细列表。
总而言之,研究通过一种多模态融合分析方法,在大量健康人群中成功识别出一个与精神分裂症遗传风险相关的、由特定脑区fALFF和GMV共同变化的复杂脑网络模式,尽管其解释的个体差异较小,但具有高度的统计学意义。
PRS-MRI linkage replication in SZ patients
这段主要讲述了研究者如何对先前识别的**PRS(多基因风险评分)-MRI(脑影像)**关联模式进行验证和特异性确认。
主要分为两个部分:
在精神分裂症患者中复制PRS-MRI关联 (PRS-MRI linkage replication in SZ patients):
- 方法:研究者将先前在健康人群中识别出的PRS相关空间脑图谱(PRS spatial maps)线性投影到290名精神分裂症患者的数据上。
- 结果:在这些患者中,PRS与该脑影像成分之间的相关性依然显著(fALFF:r = 0.35, p = 1.2e−04;GMV:r = 0.33, p = 1.4e−04)。星号表示经过FDR校正。
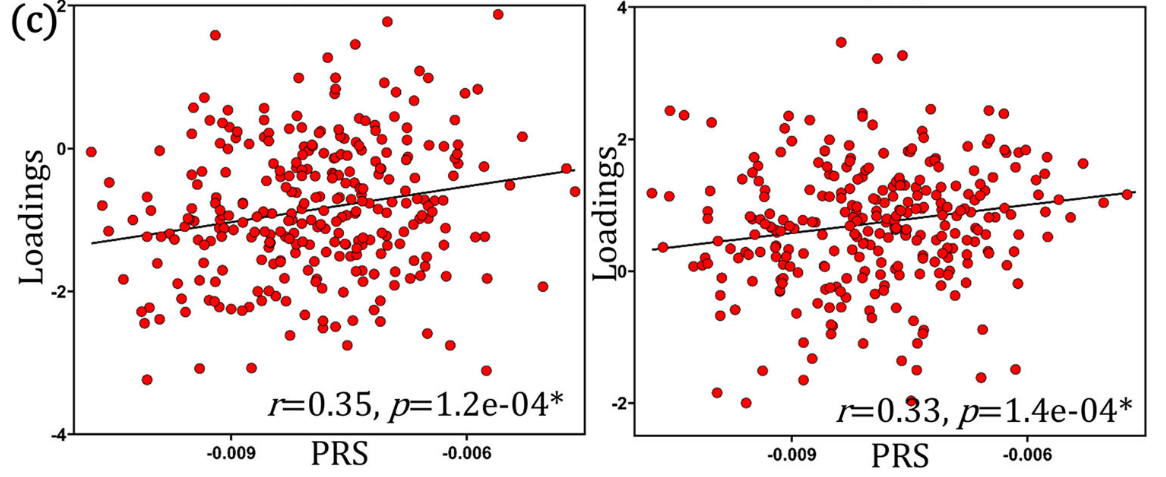
- 结论:这意味着PRS与该特定脑模式之间的关联能够在一个独立的精神分裂症患者数据集中得到复制和验证。
确认PRS相关模式的特异性 (Specificity Confirmation):
- 目的:为了证实提取出的PRS相关脑模式是特异于PRS本身的,而不是一个随机产生的模式。
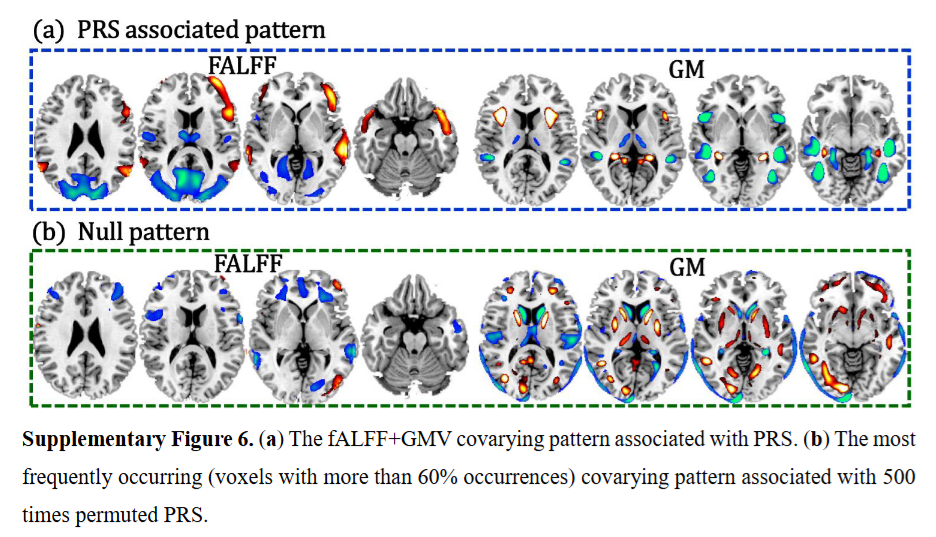
- 方法:研究者在有监督的融合分析中对PRS进行了置换检验(permuted the PRS)。这通常意味着他们打乱PRS的顺序,再进行分析,看是否还会出现类似的脑模式。
- 结果:通过置换产生的随机模式(null pattern)与先前识别出的PRS相关模式非常不相似。
- 结论:这证实了所识别的脑模式确实是与真实的PRS相关的,而非偶然或随机形成的。
总结来说,研究者成功地在一个独立的精神分裂症患者队列中复制了最初发现的PRS与特定多模态脑影像模式之间的关联。此外,通过置换检验,他们还证明了这个脑模式是特异性地与精神分裂症的PRS相关,而不是一个随机的脑活动或结构模式。
PRS-pattern consistency across PRS parameters within UKB sample
这段阐述了研究者如何在英国生物样本库 (UKB) 内部,通过改变多种参数来验证先前识别的“PRS(多基因风险评分)相关脑模式”的稳定性和一致性。
主要验证方法和结果如下:
在UKB样本内部对PRS参数进行稳健性验证:
- 方法:研究者在UKB样本的不同子集(健康的白人、所有健康被试、所有通过MRI质量控制的被试)中,使用了不同的PRS计算阈值(PSNP阈值:5.0e−08, 1.0e−04, 0.05;连锁不平衡剪枝阈值r²:< 0.1 和 < 0.2)重复了PRS引导的融合分析。
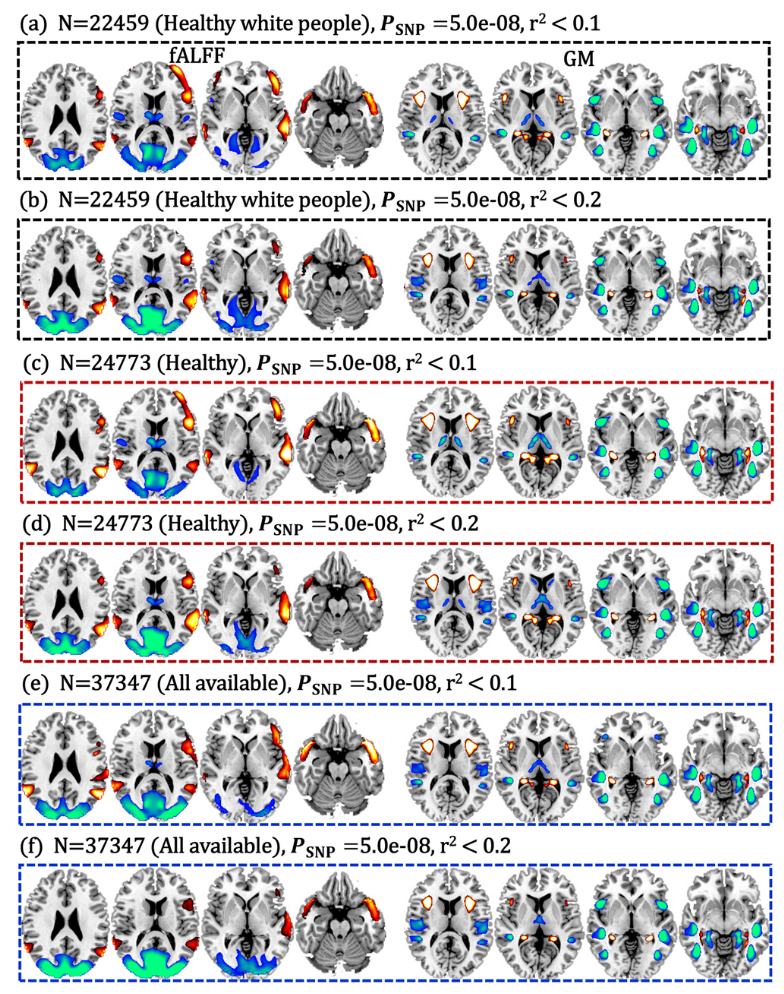
- 结果:
- 已识别的PRS相关的额颞叶模式 (frontotemporal pattern) 在UKB内部具有高度可重复性。
- 在不同的剪枝阈值和人群选择策略下,特定的fALFF模式(额中下回MIFC、颞上中回SMTC呈正相关;丘脑、后扣带皮层PCC、枕中回MOC、舌回LG呈负相关)和GMV模式(前脑岛、海马呈正相关;中脑岛、颞上中下回SMITC、梭状回FG、海马旁回呈负相关)具有超过80%的空间相似性。
- 在不同的PSNP阈值下,部分核心模式(fALFF在MIFC、SMTC的正相关,PCC、MOC的负相关;GMV在前脑岛、海马的正相关,中脑岛、海马旁回的负相关)也得到了超过50%的空间相似性验证。
使用不同版本的PRS定义进行验证:
- 方法:研究者还使用了基于更新的PGC SZ 3(包含270个位点,来自更大规模的GWAS研究)计算的PRS进行了验证。
- 结果:
- 基于PGC3和PGC2计算出的精神分裂症PRS之间高度相关。
- 使用PGC2引导的融合分析和PGC3引导的融合分析所识别出的PRS相关脑模式也非常相似(fALFF成分相似性为0.89,GMV成分相似性为0.85)。
总结来说,该研究通过在UKB样本内改变人群选择、PRS计算的PSNP阈值和剪枝阈值,以及使用来自不同GWAS研究版本(PGC2 vs PGC3)的PRS定义,均发现先前识别的核心PRS相关脑模式(特别是额颞叶区域的fALFF和GMV变化)具有高度的稳定性和可重复性。这增强了该PRS相关脑模式作为精神分裂症潜在生物标记的可靠性。
Site and motion effects on the identified PRS-pattern
这段主要评估了MRI扫描地点 (site) 和头动 (motion) 以及智商 (IQ) 这些潜在混杂因素对先前识别的“PRS(多基因风险评分)相关脑模式”的影响。
主要内容和结论如下:
- 地点效应 (Site effects):
- UKB数据来自三个不同的扫描地点(Cheadle, Reading, Newcastle)。
- 研究者分别对每个地点的数据进行了PRS引导的融合分析。
- 结果显示,在三个地点各自识别出的PRS相关额颞叶模式与整体UKB样本识别的模式之间存在高度的空间相似性(>0.70)。
- 重要前提:在最初的主要融合分析中,地点效应已经作为协变量从fALFF/GMV特征矩阵中被回归剔除了。
- 结论:因此,研究者认为扫描地点不是影响所识别PRS相关额颞叶多模态模式的主要混杂因素。
- 头动效应 (Motion effects):
- 研究者采取了多种策略来控制头动伪影的影响:
- 预处理:对fMRI数据进行尖波去除 (despiked),并回归掉包括6个头动参数在内的多种滋扰协变量。
- 被试排除:在主要分析中排除了头动过大的被试。
- 敏感性分析:选取了一个头动更小(FD < 0.2 mm, N = 13,490)的UKB子集进行PRS引导的融合分析,结果显示先前识别的PRS相关模式(额颞叶和丘脑的fALFF,以及丘脑、海马、海马旁回和颞叶的GMV)在该低头动子集中得到了复制。
- 其他证据:精神分裂症组(SZ)与健康对照组(HC)在头动上无显著差异;平均头动(mean FD)与PRS之间无显著相关性;在回归掉平均头动后,已识别成分与PRS之间的偏相关依然显著。
- 分析方法选择:当前融合分析基于对头动相对不敏感的fALFF,而非对头动更敏感的功能连接。
- 重要前提:在最初的融合分析前,平均头动也已从fALFF/GMV特征矩阵中被回归剔除。
- 结论:研究者认为微小的头动不是影响当前研究结果的主要因素。
- 研究者采取了多种策略来控制头动伪影的影响:
- 智商效应 (IQ effects):
- PRS与IQ之间的直接相关性仅为边缘显著(p = e−05,未经FDR校正)。
- 在回归掉IQ的影响后,得到的PRS相关模式与原始的PRS模式几乎相同。
- 结论:IQ不是影响所识别PRS模式的主要混杂因素。
总而言之,这段文字表明,研究者通过一系列严格的检验和控制,认为他们所识别的PRS相关脑模式对于MRI扫描地点、头动以及智商这些潜在的混杂因素是稳健的,这些因素并未对主要研究结果构成显著影响。
Group differences of PRS-pattern between SZ and HC
这段描述了研究者如何比较先前识别的PRS(多基因风险评分)相关脑模式在精神分裂症患者 (SZ) 和健康对照组 (HC) 之间的差异。
主要步骤和结果如下:
提取PRS相关脑模式特征:
- 研究者将先前识别的与PRS相关的fALFF(低频振幅分数)和GMV(灰质体积)成分,根据其Z值脑图谱,分成了“正相关网络”(Z > 0的部分)和“负相关网络”(Z < 0的部分)“掩模”(mask) 或“感兴趣区域”(Region of Interest, ROI)。
- 通过计算这些网络内fALFF或GMV的平均值,得到了四个与PRS相关的脑特征:fALFF_positive (fALFF正相关网络均值), fALFF_negative (fALFF负相关网络均值), GMV_positive (GMV正相关网络均值), 和 GMV_negative (GMV负相关网络均值)。
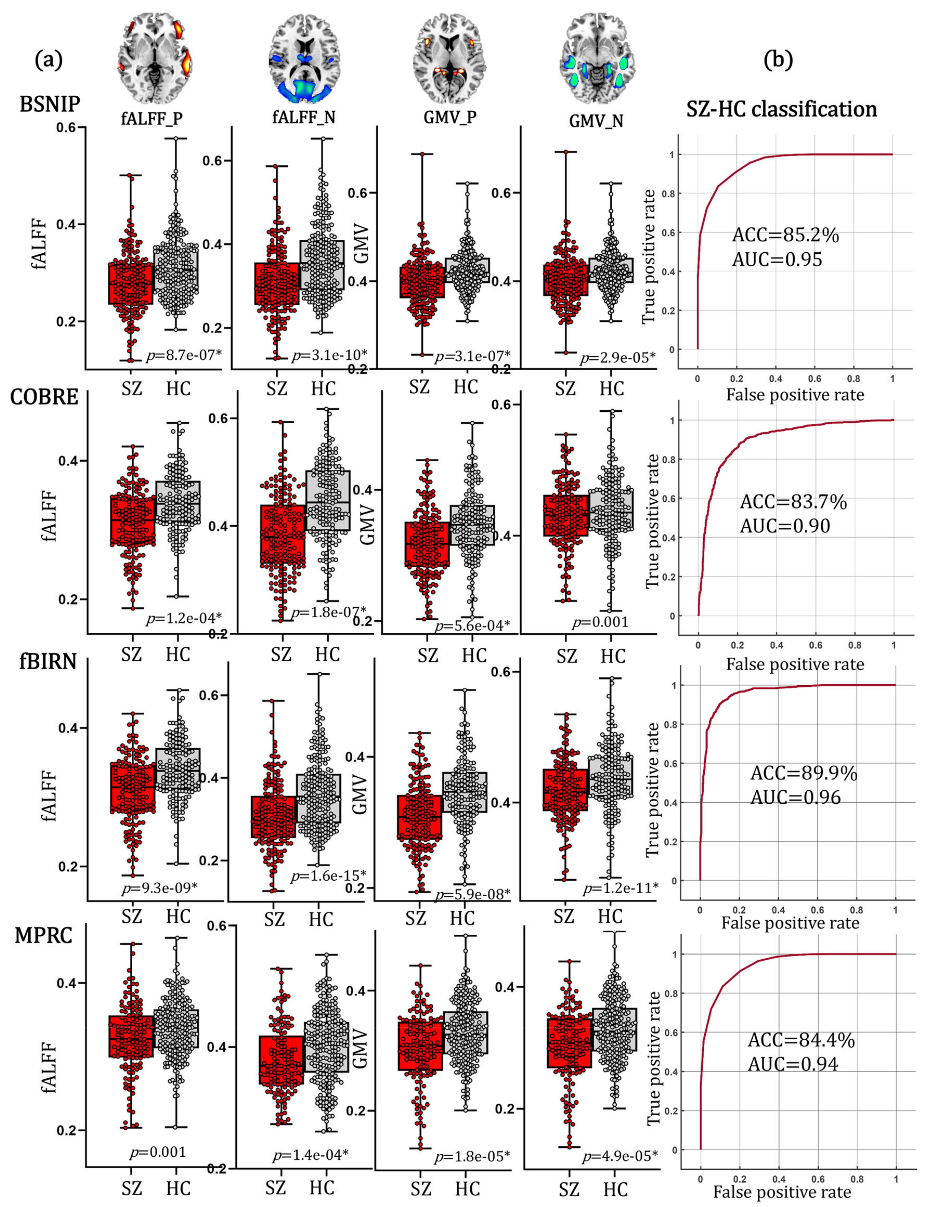
比较SZ和HC之间的组间差异:
- 使用双样本t检验来评估这四个PRS相关脑特征在SZ患者和HC之间的差异。
- 结果显示:在fMRI的正相关和负相关网络中,以及在sMRI的正相关和负相关网络中,精神分裂症患者的fALFF值和GMV值均持续显著低于健康对照组。
- 这一发现在四个独立的研究队列中都得到了验证,这些队列包括:BSNIP-1、COBRE、fBIRN和MPRC。
总结来说,研究者将先前发现的与遗传风险相关的多模态脑模式分解为四个具体的量化特征。通过比较这些特征,他们发现在四个不同的精神分裂症患者队列中,患者在所有这四个PRS相关脑特征上的值都一致且显著地低于健康对照组。这表明该PRS相关脑模式在精神分裂症患者中表现出普遍性的减弱。
Classification between SZ and HC
该研究通过使用线性支持向量机 (SVM) 方法,验证了先前识别的与精神分裂症多基因风险评分 (PRS) 相关脑网络的分类能力,用以区分精神分裂症 (SZ) 患者和健康对照组 (HC) 。
在分类特征的选择上,除了使用在PRS相关网络(fALFF/GMV的阳性和阴性网络)中提取的平均fALFF/GMV值外,研究者还纳入了在这些已识别PRS网络内分解fALFF/GMV阳性/阴性特征矩阵得到的前5个主成分 (PC) 。每个PRS相关网络因此产生6个特征(1个平均值 + 5个PC),总共形成了24个特征用于每个HC-SZ队列的分类 。加入PC的理由是前5个PC能够捕获PRS相关感兴趣区域 (ROI) 中99%的方差,而平均值仅能代表不到50%的方差 。
分类结果显示,这些PRS相关特征在4个独立的SZ队列(BSNIP-1, COBRE, fBIRN, MPRC)中均能稳定地分类SZ和HC,准确率超过83%,AUC值超过0.9 。具体表现为:BSNIP-1队列准确率85.2%,AUC为0.95;COBRE队列准确率83.7%,AUC为0.90;fBIRN队列准确率89.9%,AUC为0.96;MPRC队列准确率84.4%,AUC为0.94 。研究还发现,**第一个PC对分类的贡献最大,其次是平均值,这表明了在分类中加入额外5个PC的必要性 。**重要的是,这5个PC是从已识别的PRS相关ROI中提取的,而非全脑数据 。
为了证实分类结果的特异性,研究者使用了“无效模式”(null pattern)进行对比。结果显示,无效模式在SZ和HC之间既没有表现出组间差异,也没有区分SZ患者的能力 。这些结果证明了所识别的PRS模式在区分SZ和HC方面的特异性 。
此外,研究还评估了不同研究中心(site)作为潜在混杂因素的影响。当将不同中心视为分类目标时,分类准确率接近50%(随机水平),这表明研究中心并非当前SZ-HC分类分析中的主要混杂因素 。
Prediction of cognition and symptom for SZ
该研究进一步探讨了先前识别的四种平均PRS(多基因风险评分)相关脑特征对精神分裂症 (SZ) 患者认知和症状的预测能力 。研究人员使用COBRE队列的数据为每个认知和症状领域构建了多元线性回归模型 。随后,这些在COBRE队列上训练好的模型和相同的感兴趣区域 (ROI) 被用于预测fBIRN和BSNIP队列中SZ患者的认知及症状指标,以评估模型的泛化能力 。
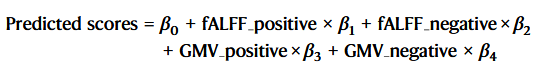
预测性能通过计算预测值与真实值之间的相关性来评估 。结果显示,这四种PRS相关脑特征成功地预测了所有三个独立SZ队列(COBRE、BSNIP-1、fBIRN)中的注意力、工作记忆、综合认知得分以及PANSS(阳性与阴性症状量表)阴性症状得分 。
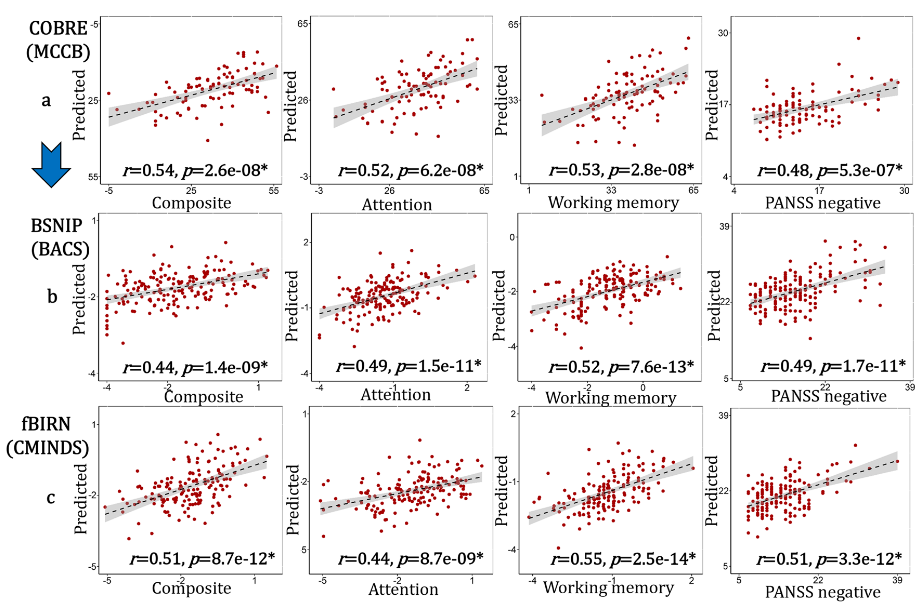
具体的Pearson相关系数 (r) 如下:
- COBRE队列:综合认知r=0.54,注意力r=0.52,工作记忆r=0.53,PANSS阴性症状r=0.48 。
- BSNIP-1队列:综合认知r=0.44,注意力r=0.49,工作记忆r=0.52,PANSS阴性症状r=0.49 。
- fBIRN队列:综合认知r=0.51,注意力r=0.44,工作记忆r=0.55,PANSS阴性症状r=0.51 。
MPRC队列因为缺乏相关的症状和认知数据,未被纳入此次预测分析 。值得注意的是,尽管三个队列使用了不同的认知评估工具(BSNIP-1使用BACS;fBIRN使用CMINDS;COBRE使用MCCB),这些PRS相关特征仍然显示出预测能力 。
Specificity of PRS pattern among psychosis and their relatives
为了检验先前识别的PRS(多基因风险评分)衍生脑模式是否也存在于其他精神病性障碍中,研究人员在BSNIP-1队列中,比较了精神分裂症 (SZ, N=178)、分裂情感性障碍 (SAD, N=134)、精神病性双相障碍 (BP, N=143)、健康对照组 (HC, N=220) 以及这三类患者的一级亲属(分别为SZR, N=162; SADR, N=149; BPR, N=142)之间四种PRS相关脑特征(fALFF正/负网络,GMV正/负网络)的差异 。所有比较在校正了地点效应后进行 。
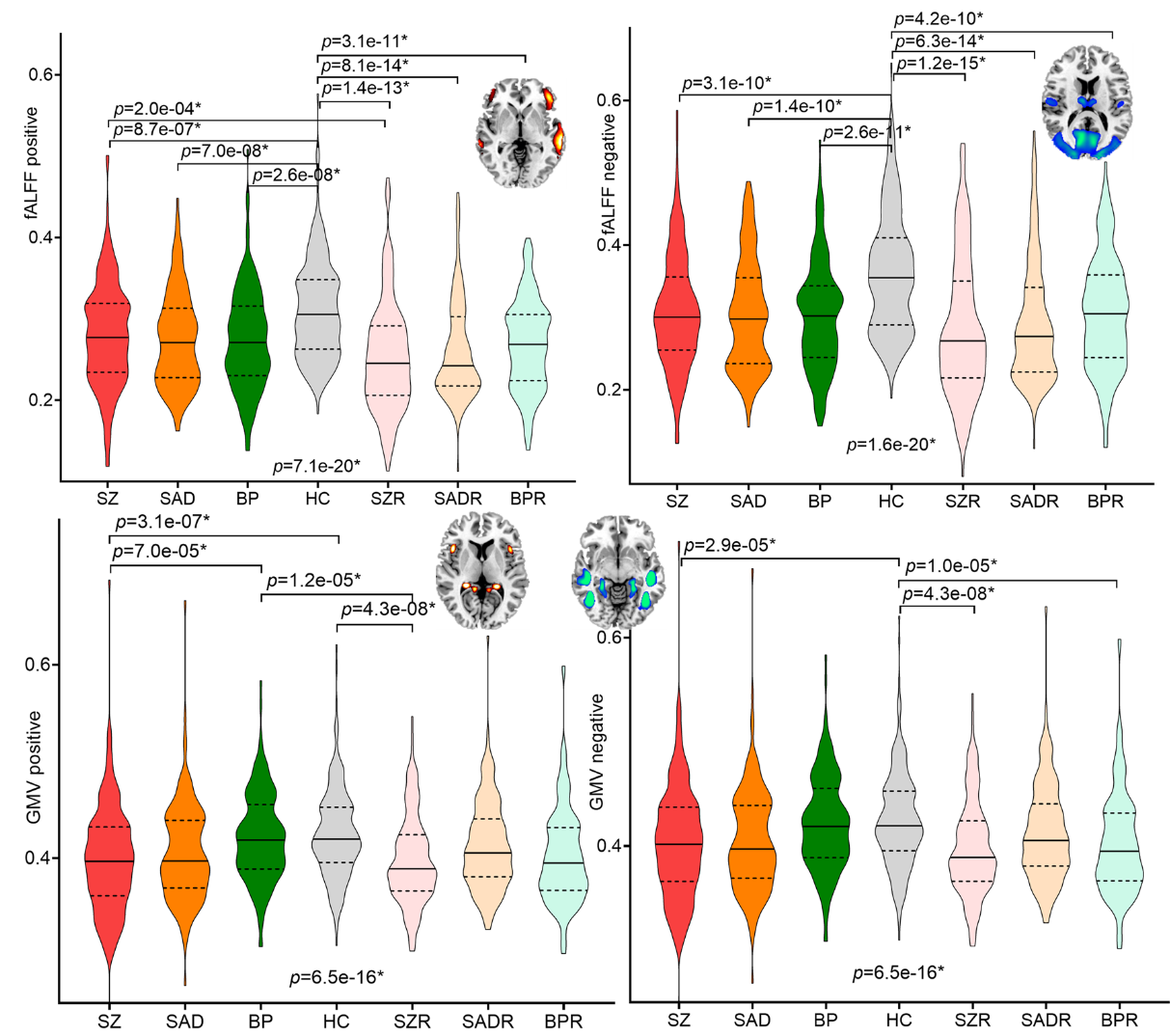
结果显示:
- 对于fALFF(功能磁共振成像特征),精神病性障碍患者组(SZ、SAD、BP)及其亲属组(SZR、SADR、BPR)在正相关和负相关网络中的fALFF值均低于健康对照组(HC) 。
- 对于sMRI(结构磁共振成像特征),与健康对照组(HC)相比,精神分裂症患者(SZ)和分裂情感性障碍患者的亲属(SADR)在正相关和负相关网络中的灰质体积(GMV)均显著减少 。
Specificity of PRS pattern among SZ, ASD, MDD, and ADHD
为了进一步检验已识别的PRS(多基因风险评分)相关脑特征的特异性,研究人员还通过双样本t检验,测试了这些特征在区分其他神经精神/心境障碍患者与健康对照组(HC)的能力 。
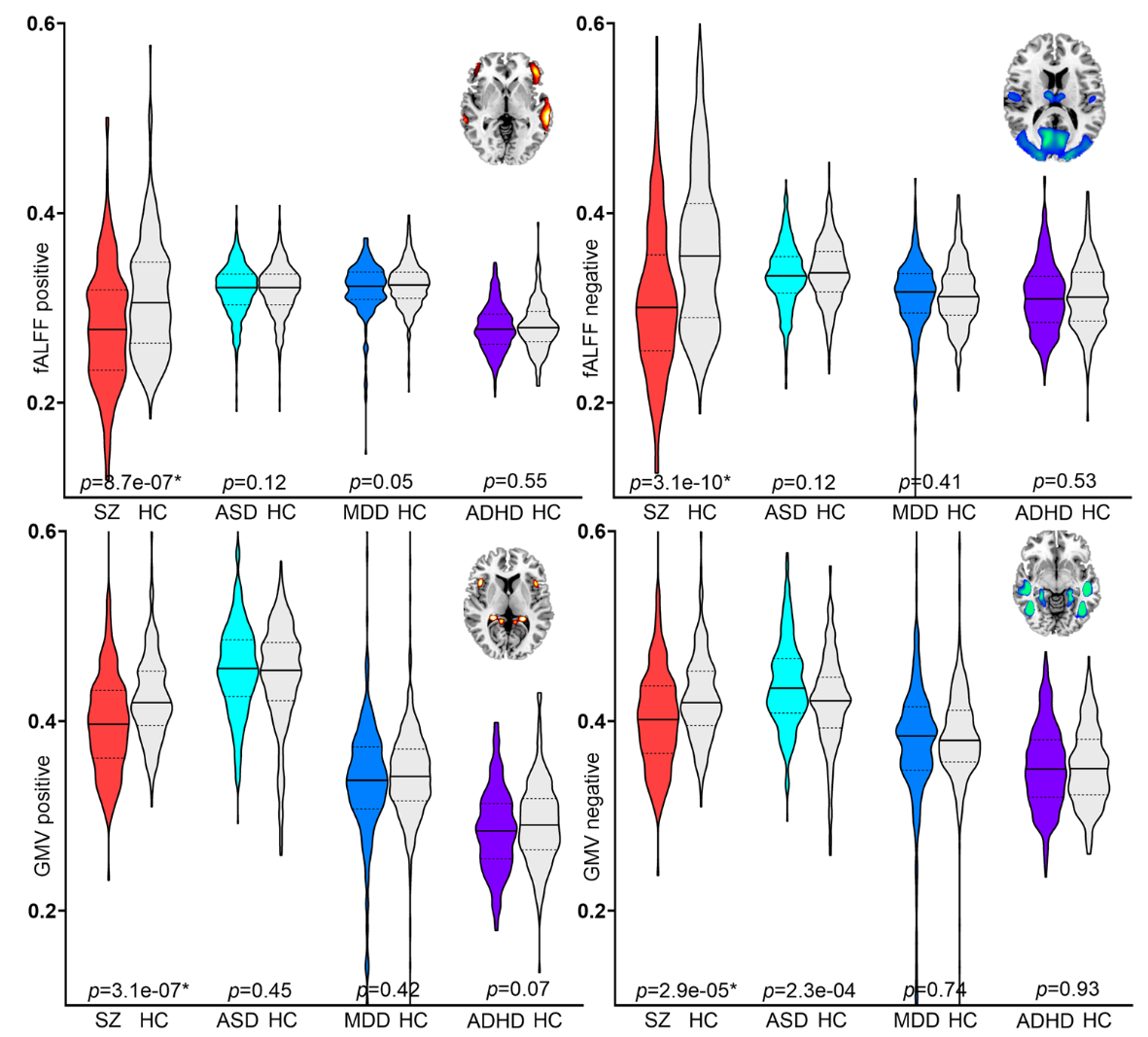
结果表明,对于患有自闭症谱系障碍(ASD, N=421)、重度抑郁症(MDD, N=260)和注意力缺陷/多动障碍(ADHD, N=346)的个体,他们所表现出的PRS相关额颞叶模式与其各自队列中的健康对照组相比,并无显著差异 。然而,该模式仅在精神分裂症(SZ)患者中显示出显著降低 。
文中还提到,关于SZ、ASD、MDD和ADHD之间的直接比较(通过方差分析ANOVA进行)可以在补充图17中找到 。
方法与材料
The discovery UKB cohort
疑惑
1. 文章里面对于获取的脑模式有两种处理方式,一种是进行线性投射求Loading,另一种是求mask求平均值,为什么要这样处理?
在神经影像分析中,选择使用“线性投影求loading”还是“直接用mask求平均值”取决于研究的具体目的和分析阶段。这两种方法有不同的应用场景和依据:
- 线性投影求Loading (成分载荷/权重):
- 目的:
- 验证或复制一个已识别的复杂空间模式(component/network map)在另一个独立样本中的表达强度,并考察这种表达强度是否与某个变量(如PRS)相关。
- 当研究者通过数据驱动的方法(如独立成分分析ICA、主成分分析PCA或文中的融合分析)识别出一个包含多个脑区、具有特定空间分布的脑网络或模式时,这个模式是一个整体。
- 为了在新的被试数据中量化这个“整体模式”的强度或“存在感”,就需要将这个已知的空间模式(通常是一个权重向量或图谱)“投影”到新被试的脑影像数据上。
- 方法:线性投影通常是通过计算新被试的脑影像数据(例如,一个向量化的全脑fALFF图)与已识别的空间模式图谱之间的点积(dot product)或其他类似的线性组合。结果得到一个单一的数值,即“loading”或“成分得分”,代表该被试在多大程度上表现出这个特定的空间模式。
- 依据/优势:
- 能够捕捉整个复杂空间模式的综合信息,而不仅仅是单个区域。
- 适用于检验一个先前发现的、由多个区域共同构成的功能或结构模式是否在新的数据中稳定存在,并与特定指标相关。
- 在文中,当他们将在UKB健康人群中识别的“PRS空间图谱”应用到独立的精神分裂症(SZ)患者队列中,并考察其与PRS的相关性时,就使用了线性投影来获得每个SZ患者在该PRS相关模式上的loading值 。
- 目的:
- 用Mask求区域平均值:
- 目的:
- 比较不同组别(如SZ vs. HC)在与PRS模式相关的特定脑区(由该模式定义)内的平均脑活动或结构是否存在差异。
- 提取简化的、具有生物学意义的特征用于分类或预测模型。
- 方法:
- 首先,基于先前识别的PRS相关脑模式图谱(通常是Z值图谱),通过设定阈值(如Z>0为正相关网络,Z<0为负相关网络)来创建二值化或多值的“掩模”(mask)或“感兴趣区域”(ROI) 。这些掩模定义了具体的脑区范围。
- 然后,对于每一个被试,计算其脑影像数据(如fALFF图或GMV图)在这些预先定义的掩模区域内的信号平均值 。
- 依据/优势:
- 简化数据和提高解释性:将复杂的全脑模式简化为少数几个关键区域的平均信号,更易于进行组间比较和生物学解释。例如,可以直接说“SZ患者在PRS正相关网络的平均fALFF值显著低于HC” 。
- 降低特征维度:对于分类和预测任务,使用少数几个区域的平均值作为特征,可以有效降低模型的复杂性,减少过拟合的风险,并可能提高模型的泛化能力 。文中明确提到,提取了4个PRS相关的脑特征(fALFF_positive, fALFF_negative, GMV_positive, GMV_negative)用于后续的组间比较、分类和预测分析 。
- 特异性分析:允许研究者更细致地考察模式中不同部分(如正相关部分和负相关部分)的效应。
- 目的:
总结与依据选择:
- 当需要评估一个复杂的、数据驱动发现的整体脑模式在个体中的表达强度,并将其与外部变量关联时(尤其是在验证/复制阶段),倾向于使用“线性投影求loading”。这能反映个体对整个模式的“贡献”或“符合程度”。
- 当需要进行组间比较,或者为机器学习模型(分类、预测)提取更稳定、更易解释、维度更低的特征时,且这些特征应基于先前发现的模式所指示的关键区域时,倾向于使用“mask求区域平均值”。这种方法将关注点从整个模式的抽象表达转移到模式中具体解剖或功能区域的量化指标上。
研究者会根据研究的不同阶段和具体科学问题来选择合适的方法。在本文中,两种方法都被恰当地用于回答不同的研究问题:线性投影用于验证PRS与脑模式的关联在患者中的可重复性 ;而基于mask的区域平均值则用于更细致地比较SZ与HC在这些PRS定义区域的差异 ,并作为特征进行分类和预测 。